単一細胞レベルでのmRNA発現解析の定量評価
Quantitative Assessment of mRNA Expression Analysis at the Single-Cell Level
米川 侑希
*鈴木 幸栄
*黒澤 信幸
**磯部 正治
**Yuuki YONEKAWA Koei SUZUKI Nobuyuki KUROSAWA Masaharu ISOBE
要 旨
_________________________________________________
近年,個々の細胞の機能や性質を知るための単一細胞を対象としたmRNA (messenger ribonucleic acid: 伝令RNA) 発現解析が注目されている.しかし,濃度の測定精度が確保さ れた核酸を用いた評価は十分に行われておらず,単一細胞のmRNA発現解析の精度は明確で はない.そこで,濃度が既知の認証標準物質である定量解析用リボ核酸 (RNA) 水溶液を用 いて,mRNA発現量の定量可能な範囲の特定を行った.RNA抽出とcDNA (complementary deoxyribonucleic acid: 相補的DNA) 合成は,独自開発中の自動cDNA合成装置で行い,RNA から合成したcDNAをリアルタイムPCR (polymerase chain reaction: ポリメラーゼ連鎖反応) 装 置で検出し,検出対象RNAの量を定量化した.その結果,単一細胞レベルであればmRNAは
3~107コピーと少ないコピー数から定量的に解析可能であることが確認された.この定量評
価により,認証標準物質を用いて,mRNA発現解析の精度の評価ができることを示すことが できた.
ABSTRACT _________________________________________________
Recently, messenger ribonucleic acid (mRNA) expression analysis at the single-cell level has attracted attention for investigating the function and properties of individual cells. However, little has been reported on the accuracy of gene expression analysis for a single cell. Therefore, we investigated the dynamic range of this analysis using certified reference material (CRM) containing a defined concentration of RNA. The methods of RNA capture and complementary deoxyribonucleic acid (cDNA) synthesis were both performed with our automated cDNA synthesizer and the quantity of copies of cDNA was measured by real-time polymerase chain reaction (PCR).
Results showed that RNA molecules from 3 to 1.0 × 107 copies at the single-cell level can be
accurately quantified using our system. This demonstrates that expression analysis of very low copy numbers of mRNA molecules at the single-cell level can be performed. In this work, we demonstrate that the use of CRM enables us to evaluate the accuracy of mRNA expression analysis.
* 研究開発本部 リコー未来技術研究所 バイオメディカル研究室
Biomedical Research Department, Ricoh Institute of Future Technology, Research and Development Division
** 富山大学大学院 理工学研究部
1.
背景と目的
1-1
mRNA発現解析
細胞内には,デオキシリボ核酸 (deoxyribonucleic acid: DNA) とリボ核酸 (ribonucleic acid: RNA) があ り,これらは生命活動の維持に重要な働きをする1).
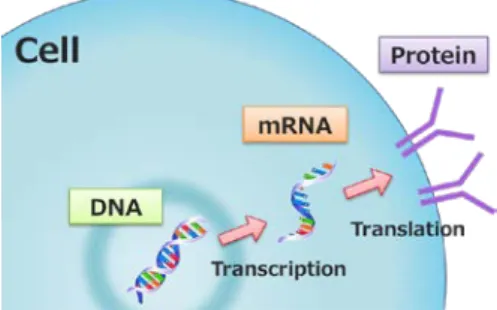
遺伝子は遺伝情報の単位で,遺伝子発現とはDNA の遺伝情報がmRNA (messenger RNA: 伝令RNA) に 転写され,mRNAの遺伝情報を翻訳してタンパク質 を合成する過程のことをいう (Fig. 1) 2).細胞の構 造や機能を決める遺伝子の発現情報解析は,タンパ ク質を解析するよりも高感度に評価が行えることか ら,微量の細胞内の状態把握に適している.さらに mRNAの発現量は日々変化するため,細胞状態の変 化を検出するのにも役立つ. 一般的に,mRNA発現解析はリアルタイムPCR (polymerase chain reaction: ポリメラーゼ連鎖反応) 装置を用いて行う.本装置では,リファレンスとな る材料を用いて検量線を作成し,測定したいmRNA 発現量を定量することができる.リアルタイムPCR 装置の詳細は,次章で説明する.
Fig. 1 The process of gene expression involves two steps. The first is transcription, where the DNA sequence of a gene is copied into mRNA. mRNA carries genetic information for making protein. The second step is translation. In this step, the sequence of mRNA is translated to the amino acid sequence in protein.
1-2
単一細胞のmRNA発現解析
細胞は一つひとつが異なった役割を担っており, 同一種の細胞でもmRNA発現量は異なっている.し かし,従来のmRNA発現解析は細胞集団を対象とし ていたため,個々の細胞のmRNA発現量は,平均化 されて把握できなかった (Fig. 2) 3).そこで近年, 個々の細胞の状態を把握できる単一細胞のmRNA発 現解析が注目されている4).個々の細胞を解析する ことで,細胞間のmRNA発現量の不均一性を把握で きるため,細胞培養の周期や品質の評価をより正確 に 行 え る . 特 に 人 工 多 能 性 幹 細 胞 (induced pluripotent stem cell: iPS細胞) 由来の細胞移植におい ては,iPS細胞が対象組織の細胞に正しく分化誘導 されたかどうかの評価に用いられており,再生医療 分野で有用である5).また,がん組織内の細胞を個々で解析し,腫瘍内不均一性を調べることは,が んの基礎研究や治療方針の決定に役立つ.
しかし,単一の細胞に含まれる総RNA (total RNA) 量は約0.01 ngとされ,その中でmRNAが占める割合 は1~2%であり,わずか約1.0×10-4 ngしか存在しな
い6).これは,単一細胞のmRNA発現解析では,数
コピーのmRNAを定量化する必要があることを意味 する.
Fig. 2 mRNA expression analysis at the cell-population and single-cell level. (a) Analysis at the cell population provides an averaged mRNA expression profile that is insensitive to variation between individual cells. (b) In contrast, single-cell analysis enables us to detect mRNA expression heterogeneity among cells.
1-3
mRNA発現解析の現状と課題
リアルタイムPCR装置を用いたmRNA発現解析で は,検量線を作成してmRNA発現量を定量する.そ の際にリファレンスとするサンプルは,一般的に核 酸濃度を測定し,濃度を定める.しかし,核酸濃度 を測定する装置の測定妥当性の検討は十分ではない. 代表的な核酸濃度の測定方法として,吸光度から核 酸濃度を測定する方法がある.本測定は,核酸の塩 基が260 nm付近の紫外線を吸収する性質を利用する. 核酸濃度ρ
B [ng/μL]は式(1)に示すように,測定した 吸光度𝐴𝐴
,波長に依存する吸光係数𝜀𝜀
[ng-cm/μL], 光路長𝑙𝑙
[cm]を用いて算出される.吸光係数𝜀𝜀
は核 酸の配列や塩基数によって異なるにもかかわらず, 一般的に一定値として計算される7).したがって, 算出される核酸濃度は吸光係数𝜀𝜀
による誤差が含ま れ,正確な濃度は不確かである.リアルタイムPCR 装置で定量評価を行う際は,既知濃度で測定誤差が 明確な核酸で検量線を引く必要がある.ρ
B = (𝐴𝐴 ∗ ε) / 𝑙𝑙(1) 近年,再生医療の発展やバイオテクノロジーの産 業化の進展に伴い,国際標準化委員会のISO/TC 276 で核酸の定量法の国際規格が提案され,核酸濃度の 定量に関する標準化の必要性が高まりつつある8).
アメリカでは,2005年にMAQC (MicroArray Quality Control) プロジェクトでマイクロアレイを用いた RNAの解析方法や品質管理の標準化に取り組んだ. その際に,RNAの外部標準試料 (Universal Human Reference RNA: UHRR) を用いた評価を実施した9).
しかし,RNAの外部標準試料は複数種のRNAが混 合したtotal RNAの濃度しか定義されておらず,リ アルタイムPCRのような個々のmRNA発現量の定量 には用いることはできない.また,ERCC (External RNA Control Consortium) でも外部標準試料が作製 されたが,濃度の測定方法は明らかではなく,相対 的な濃度比較用のため,リアルタイムPCRでの定量 評価に適用できるかは不明である10). 国内では,2013年に国立研究開発法人産業技術研 究 所 が 認証 標 準物 質 として 定 量 解析 用 リボ 核酸 (RNA) 水溶液を開発した11).本標準物質は,自然 界に存在するゲノム配列などと類似性が低い人工的 な塩基配列を有するRNAで構成され,ISO GUIDE 34: 2009およびISO/IEC 17025: 2005に適合するマネ ジメントシステムに基づき生産されている.RNA の濃度は同位体希釈質量分析法と誘導結合プラズマ 質量分析法 (ICP-MS) を用いて国際単位系 (SI) に トレーサブルに定量されて値付けされている.本標 準物質を用いてmRNA発現解析の定量評価が行われ ているが,低濃度のmRNA発現解析に関しては報告 例がない12). そこで,我々は本標準物質を用いて,リアルタイ ムPCR装置で定量可能なmRNAの濃度範囲を特定す る方法を検討した.
2.
mRNA発現解析の定量評価
2-1
定量評価方法
2-1-1 サンプルの調製 mRNA発現解析の定量評価には,1章で示した外 部標準試料 (UHRR) と定量解析用リボ核酸水溶液 NMIJ CRM 6204-1 No.032 (RNA1000-B) をサンプル として用いた.UHRRはヒトの10種類の細胞株から プールされたtotal RNAの混合物で,RNAの総質量 が定められている.RNA1000-Bは,塩基長や質量 濃度,物質量濃度などが定められている (Table 1). ま た , RNA1000-B は 塩 基 配 列 の 片 側 の 末 端 に mRNA特有のポリA配列が付加されている.そこで, 本評価ではRNA1000-BをmRNAのモデルサンプル として用いた. 本評価では,検出可能なmRNAのコピー数を定量 するため,サンプルをUHRRとRNA1000-Bの混合物, ターゲットを既知濃度のRNA (RNA1000-B) とした. UHRRの質量は,単一細胞,100細胞,10万細胞を 想定して,0.01,1,1000 ngとした.検出するRNA(RNA 1000-B) のコピー数は,3,6,13,25,50, 100,500,1000,1.0×104,1.0×105,1.0×106, 1.0×107コピーとした6).それぞれの量を変えるこ とで,夾雑物となるtotal RNAの割合に対して検出 可能なmRNAの濃度範囲を定量できる条件とした. なお,コピー数
𝑥𝑥
は式(2)に示すように,アボガド ロ 定 数 NA [mol-1] , 物 質 量 濃 度𝑐𝑐
[mol/μL] , 液 量𝑉𝑉
[μL]を用いて算出される.𝑥𝑥 = N
A∗ 𝑐𝑐 ∗ 𝑉𝑉
(2)Table 1 Concentration of RNA1000-B. Reference value uncertainty Expanded Mass concentration 59.5 ng/μL 5.3 ng/μL Substance concentration 0.179 pmol/μL 0.018 pmol/μL
2-1-2 mRNA抽出 ・ cDNA合成
mRNA発現解析では,まず前処理として,サンプ ルからmRNAを抽出し,抽出したmRNAを鋳型に DNA合成を行う逆転写プロセスを行う (Fig. 3). mRNAは微量で不安定な物質のため,解析する際は, 逆転写酵素を用いてcDNA (complementary DNA: 相 補的DNA) 合成をしてから解析する.
本評価では,ポリA配列が付加されたRNAおよび mRNAの抽出と cDNA合成を,独自開発中の自動 cDNA合成装置を用いて行った.自動cDNA合成装 置 は 懸 垂 液 滴 ア レ イ 式 磁 気 ビ ー ズ 反 応 法 (MAGnetic beads Reaction through Arrayed Hanging Drops: MAGrahd法) を応用した装置で,反応用プ レート上に反応液滴を分注し,mRNA抽出とcDNA 合成を自動で行うことができる (Fig. 4) 13). 反応としては,ポリA配列をもつRNA (mRNA) を捕捉するポリT配列を表面に付加したオリゴdT磁 気ビーズを用いてmRNA抽出を行い,磁気ビーズ上 で mRNA 全 長 の cDNA 合 成 を 行 う . 試 薬 に は , mRNA抽出にDynabeads mRNA DIRECT Purification Kit, cDNA合成にSuperScriptⅢ Reverse Transcriptase,
RNaseOUT Recombinant Ribonuclease Inhibitor, dNTP Mixを用いた. 装置の特徴としては,微量の液滴で反応を行うた め,低濃度のmRNAを高効率で抽出可能なこと,最 大144サンプルが同時に処理可能でハイスループッ トなことがある. しかし,検出可能なmRNAのコピー数は定量化さ れていないため,今回の評価で夾雑物中から検出可 能なmRNAのコピー数を明らかにした.なお,本評 価では初期のサンプル量および反応液量を3 μLで実 施した.
Fig. 3 Workflow of mRNA expression analysis.
Fig. 4 Automated cDNA synthesizer based on MAGrahd method (currently under development). (a) Image of cDNA synthesizer. (b) Schematic illustration of reaction plate for cDNA synthesis.
2-1-3 DNA増幅 ・ 検出
自動cDNA合成装置で合成したcDNAは,リアル タイムPCR装置を用いたインターカレーター法で検 出した.一般的に,cDNAは微量であるため,検出
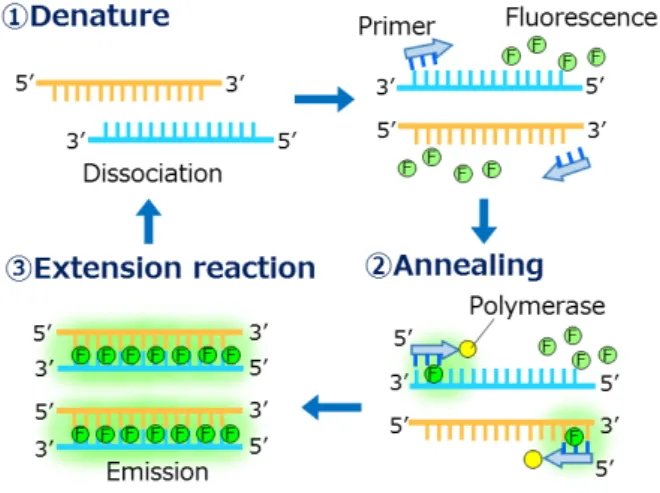
可能な量までDNAを増幅して検出する.PCR法は, 熱変性→アニーリング→伸長反応の3ステップを1サ イクルとし,このサイクルを繰り返すことでDNA を増幅する方法である.まず熱変性を行い,熱を加 えて二本鎖DNAを一本鎖DNAに解離させる14).次 にアニーリングを行い,一本鎖DNAの相補的な部 分にプライマーを結合させる.プライマーは検出し たいDNAの特定部分に特異的に結合する塩基配列 を もつ 合成オ リゴ DNAで , 温度 を下げ ると鋳 型 DNAに 結 合す る.最 後の伸 長反 応では ,プラ イ マーを起点としてDNAポリメラーゼ酵素によって DNA合成が起こり,任意の領域のDNAを二本鎖に 合成する.このサイクルを繰り返すと,1コピーの DNAは理論的には指数関数的に増幅される15). リアルタイムPCR法は,二本鎖DNAに結合する ことで蛍光を発する試薬を用い,その蛍光量を測定 することで増幅していくDNA量を算出する方法で ある (Fig. 5) 15).リアルタイムPCR装置では,主に 以下の3つの結果を図で表すことが可能である. (1) 増幅曲線 1サイクルごとのDNA量を蛍光のシグナル強度 で測定し,横軸をサイクル数,縦軸を蛍光の シグナル強度でプロットした図である.増幅 曲線は,増幅産物 (DNA) 量が蛍光検出できる 量になると立ち上がり,DNAポリメラーゼ酵 素の活性の低下やプライマーの減少などで反 応が進まなくなるとプラトーに達する過程を 示す.ターゲットの初期のDNA量が多いほど 増幅産物量は検出可能な強度に早く達する. 理論的にはターゲットの初期のDNA量が2倍に なると,立ち上がりは1サイクル早くなる. (2) 検量線 増幅曲線で蛍光のシグナル強度が立ち上がり 始めた後,ある一定のシグナル強度の時のサ イクル数をCt (Threshold Cycle) 値とし,横軸を 濃度,縦軸をCt 値でプロットした図である. Ct値とターゲットの初期のDNA量の間には直 線関係があるため,検量線が作成可能である. 検量線の傾きから反応効率の算出,検量線に 未知濃度のターゲットのCt値をプロットする ことで濃度の定量ができる. (3) 融解曲線 増幅後,PCR反応液の温度を徐々に上げ,熱変 性で二本鎖DNAが一本鎖に解離する過程のシ グ ナ ル 強 度 を 測 定 し , 横 軸 を 温 度 , 縦 軸 を -ΔF/ΔT(蛍光変化/温度変化)でプロットし た図である16).融解曲線は,二本鎖DNAが解 離し,シグナル強度が急激に低下する温度が ピークとして示される.シグナル強度の変化 のピークが複数回ある場合,ターゲット以外 の産物が増幅していると判断できる. 本評価では,サンプルをUHRRとRNA1000-Bの混 合物,ターゲットをtotal RNA中に含まれる既知濃 度のRNA (RNA1000-B) とした.そして,RNA1000-Bを用いて合成したcDNAを鋳型として,リアルタ イ ム PCR 装 置 に よ る 検 出 を 行 っ た . 試 薬 は SsoAdvanced Universal SYBR Green Supermix,プラ イマーは増幅長が150 bpのものを,リアルタイム PCR装置はQuant Studio 7 Flex Real-Time PCR System を用いてサンプル以外の試薬組成や反応条件は全て 統一した.なお,ターゲットのRNA1000-Bは他の 遺伝子中には含まれない人工配列を有しており,夾 雑物となるUHRR由来のcDNAからは増幅されない.
2-2
定量評価結果
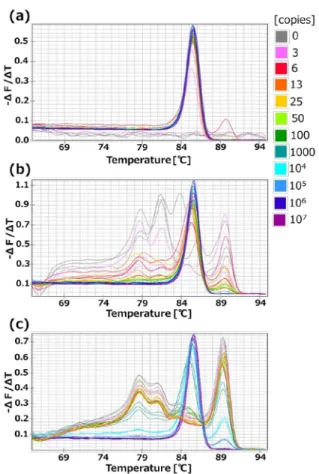
Fig. 6に,Ct値の測定結果を示す.サンプルの細 胞 数 ( total RNA の 質 量 ) 別 に , タ ー ゲ ッ ト の RNA1000-Bのコピー数に対するCt値をプロットし ている.Ct値はリアルタイムPCR装置が定めたシグ ナル強度をThresholdと増幅曲線の交点のサイクル 数とした.単一細胞レベルのtotal RNAをサンプル に用いた結果では,低濃度側でCt値のバラツキが あったが,Ct値は直線上にプロットされた.一方, 100細胞や10万細胞レベルのtotal RNAをサンプルに 用いた結果では,ターゲットのRNA1000-Bが低濃 度になるにつれてCt値の変化がなくなった.この原 因を確認するために,融解曲線を確認した.Fig. 6 Determination of the limit of quantification for detecting RNA1000-B in total RNA from different number of cells. Fig. 7に融解曲線データを示す.サンプルの細胞 数(UHRRの質量)別の融解曲線の結果を表してい る.Fig. 7より,サンプルが単一細胞レベルの時, 熱変性によるシグナル強度の変化が1回で,ター ゲットのみを特異的に検出できることがわかった. し た が って , 単一 細 胞レベ ル の 夾雑 物 中か ら, RNA1000-Bは3から1.0×107コピーの範囲で検出可 能なことが確認された.一方,サンプルが100細胞 と10万細胞レベルの時は,夾雑物となるUHRRの濃 度が高く,かつターゲットのRNA1000-Bの濃度が 低くなるにつれ,ピークの数が増えて別のピークが 大きくなった.融解曲線のピークの数が増えるにつ れてFig. 6のCt値の検量線の傾きは緩やかになり, 別のピークが大きくなるにつれてCt値の変化はなく なった.この原因は,ターゲット以外の産物の増幅 がシグナルとして検出され,その強度を含めたCt値 になったことで,定量ができなかったことを示して いる.
Fig. 7 Melting curve analysis at (a) single-cell level, (b) 100 cells level, and (c) 105 cells level. The
shape of the curve varies with different DNA sequences, enabling us to check the presence of nonspecific amplification products.
2-3
考察
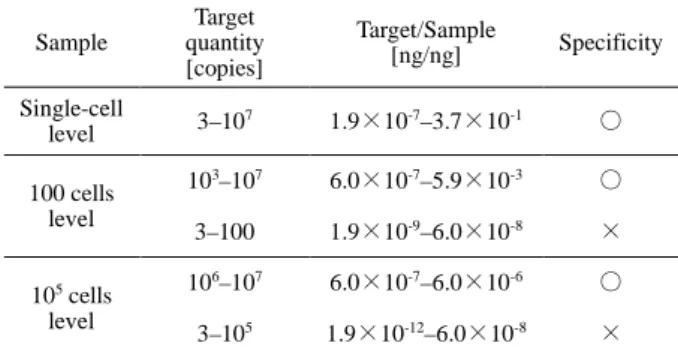
単一細胞レベルのtotal RNAからRNA1000-Bを検 出した際,Fig. 6とFig. 7より,直線上にCt値がプ ロットされ,非特異的な産物も検出されなかった. しかし,低濃度側はCt値のバラツキが大きかった. この原因は,RNA1000-Bを段階希釈する過程,低 濃度RNAの分注で生じるバラツキによるもので100 コピー以下では許容されるバラツキと考えられる.Table 2に定量評価結果をまとめた.融解曲線の結 果を基に,ターゲットのRNA1000-Bのみを検出し た条件を○,ターゲット以外の産物を検出した条件 を×で表している.この結果より,サンプルが単一 細胞レベルの時,RNA1000-Bは全ての条件の3から 1.0×107コピーと広いダイナミックレンジで検出可 能なことが示された.しかし,100細胞レベルでは 100 コ ピ ー 以 下 , 10 万 細 胞 レ ベ ル で は 1.0 × 105コ ピー以下のRNA1000-Bの時にターゲット以外の産 物が検出され,他の産物の量も含めてシグナル強度 が検出された.この結果を質量比に変換すると, ターゲット対サンプルが1.9×10-7以上であればター ゲットのみを検出可能なことが確認された. 以上より,単一細胞レベルのtotal RNAから低濃 度の3コピーのRNA1000-Bを自動cDNA合成装置で 抽出し,リアルタイムPCR装置で検出可能なことが 定量的に示された.また,トレーサビリティの確保 された材料を用いて,検出精度をターゲット対サン プ ル の 質 量 比 が 示 せ た . な お , RNA1000-B は , mRNA特有のポリA配列をもっているため,本評価 の方法は実際の実験系における数コピーのmRNA検 出に適応可能と考える.
Table 2 Summary of quantitative assessment. Sample Target quantity [copies] Target/Sample [ng/ng] Specificity Single-cell level 3–107 1.9×10-7–3.7×10-1 ○ 100 cells level 103–107 6.0×10-7–5.9×10-3 ○ 3–100 1.9×10-9–6.0×10-8 × 105 cells level 106–107 6.0×10-7–6.0×10-6 ○ 3–105 1.9×10-12–6.0×10-8 ×
3.
単一細胞レベルでのmRNA発現解析
2章より,自動cDNA合成装置でRNA抽出,cDNA 合成を行った結果,単一細胞レベルのtotal RNAか ら3コピー以上のRNA1000-BをリアルタイムPCR装 置で検出可能なことが明らかになった.そこで,ア プリケーションの検討例として,皮膚の細胞の分化 状態を評価することを想定し,単一細胞レベルの細 胞溶解液においてmRNA発現解析を実施した.3-1
方法
mRNA発現解析を行う細胞のサンプルに,表皮の 角化細胞PR3D-HPEK-50を未分化状態で培養した細 胞を用いた.表皮の角化細胞は,未分化度や分化度 で発現が異なり,状態別で特異的に発現するmRNA が発見されている17).表皮は層になっており,基底 層で細胞分裂することで角化細胞が生成される.そ して,分化が進むと古くなり,角質になる.つまり, 細胞は基底層の時は未分化状態となる.本実験では, ほとんど全ての細胞で一定量発現するリファレンス のハウスキーピング遺伝子GAPDHと,表皮の角化細 胞が未分化状態の時に特異的に発現するマーカー18) KERATIN 14の発現解析を行った.本実験はサンプ ルが未分化状態で培養した細胞のため,KERATIN 14の発現量は多いことが想定される. サンプルは10万個の細胞を溶解液で溶かした後, 100細胞,単一細胞レベルに希釈したものを用いた. mRNA発現解析は,2章と同様の反応試薬を用い て,自動cDNA合成装置でmRNA抽出とcDNA合成 を実施し,リアルタイムPCR装置で検出した.なお, プライマーは,GAPDHは増幅長が138 bp,KERATIN 14は増幅長が126 bpのものを使用した.3-2
結果
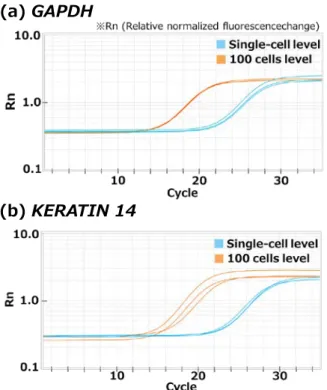
Fig. 8に増幅曲線データ,Fig. 9に融解曲線データ を示す.ハウスキーピング遺伝子GAPDH,表皮の 角化細胞が未分化の時のマーカーKERATIN 14の発 現解析の結果,単一細胞,100細胞レベルのサンプ ルの時,ともにDNAの増幅が確認された.また, 融解曲線からターゲットのみが検出されていること が確認された.よって,細胞の状態を表すマーカー を自動cDNA合成装置で抽出し,リアルタイムPCR 装置でmRNA発現解析を行えることが検証された.Fig. 8 Amplification curve of (a) GAPDH and (b)
KERATIN 14 at single-cell and 100 cells level.
Fig. 9 Melting curve analysis for (a) GAPDH and (b)
KERATIN 14 at single-cell level and 100 cells
level. Note that a single peak was observed for each reaction, indicating that a specific sequence was amplified by PCR.
3-3
考察
本実験で,開発中の自動cDNA合成装置を用いた mRNA抽出とcDNA合成,リアルタイムPCR装置を 用 い た DNA 増 幅 で , 細 胞 特 異 的 な マ ー カ ー の mRNA発現解析が単一細胞レベルのサンプルで実証 された.PCRにおいて,サンプルの細胞数が100倍 になると,理論値では立ち上がりサイクル数は約 6.7サイクル異なる.Fig. 8より,細胞数の差に対す る曲線の立ち上がりサイクル数の差は,GAPDHで 6サイクル,KERATIN 14で7サイクル程度であり, 理論値に近い値となった.よって,本手法で単一細 胞レベルのサンプルを用いても100細胞レベルのサ ンプルの時と同等の効率でmRNA捕捉,cDNA合成 が行える可能性が示せた. 細胞特異的なmRNAマーカーの発現解析を行うこ とで,細胞の分化やがん化などの状態を調べること ができる.よって,そのような用途における本手法 の適用可能性が示せたと考える.ただし,本実験は 発現量の多いmRNAをターゲットとしているため, 今後は発現量の少ないmRNAをターゲットとした発 現解析の検討も必要と考える.4.
まとめ
トレーサビリティの確保された既知濃度の認証標 準物質を用いて,リアルタイムPCR装置で検出可能 なRNA量の定量評価を実施した.評価は,開発中 の自動cDNA合成装置でRNA抽出・cDNA合成をし た後,リアルタイムPCR装置でDNA増幅をして, ターゲットのRNAを検出した.その結果,本手法 は単一細胞レベルのtotal RNAから,3から1.0×107 コ ピ ー と低 濃 度か ら 広いダ イ ナ ミッ ク レン ジで RNAが検出可能なことを定量的に確認した.この 評価では,これまで取り組まれていなかったトレー サビリティの確保された材料を用いてリアルタイム PCR装置で検出可能なRNA量を定量した.そして, 従来よりも低濃度のRNAの定量に成功し,精度を 初めて明らかにした.評価に用いたRNA1000-Bは,(a) GAPDH
(b) KERATIN 14
(a) GAPDH
(b) KERATIN 14
mRNA特有のポリA配列をもっているため,本評価 の方法は実際の実験系における数コピーのmRNA検 出に適応可能と考える.また,単一細胞レベルの細 胞溶解液をサンプルとし,細胞の状態を表すマー カーのmRNA発現解析が行えることも検証された. 今後,再生医療の実用化が進み,加工した細胞の 移植機会の増加が予想される.再生医療の実用化に おけるレギュラトリーサイエンスの課題として,移 植細胞の品質や安全性の確保がある19).また,自己 iPS細胞由来網膜色素上皮細胞に関する評価指標と して,未分化細胞が混在していないことをPCRで確 認できることが示されている20).よって,PCRのト レーサビリティの管理が必要となると考える.細胞 加工や組織形成をする際の工程検査として,トレー サビリティが管理されたPCRを用いて単一細胞レベ ルでの評価が行えれば,高品質の細胞を確実に培養 して安全で信頼性の高い移植も実現可能と考える. 我々はバイオ分野への貢献を目指し,他分野で培っ てきた品質管理に対する考えを基に定量評価の必要 性や測定妥当性の重要性を今後も広めていきたい. 参考文献 _________________________________ 1) 松村明: 大辞林, 第3版, 三省堂 (2006). 2) 井出利憲: 分子生物学講義中継 Part 1, p. 151, 羊土社 (2002). 3) 細野直哉: 幹細胞研究における単一細胞遺伝子 発現解析, 細胞, Vol. 44, No. 9, pp. 398–402 (2012). 4) A. Ståhlberg, M. Bengtsson: Single-cell gene
expression profiling using reverse transcription quantitative real-time PCR, Methods, Vol. 50, No. 4, pp. 282-288 (2010).
5) 杉田直: iPS細胞の網膜再生医療-網膜変性疾患 への臨床応用, 日本臨床免疫学会会誌, Vol. 38, No. 2, pp. 79–85 (2015).
6) G. S. Ginsburg, H. F. Willard: Genomic and Personalized Medicine, Academic Press, p. 159 (2008). 7) 柴山祥枝: 核酸標準物質の現状と動向, 産総研
計量標準報告, Vol. 8, No. 1, pp. 141–153 (2010).
8) 日置達男, 柳田豊: 標準化部会国際標準化関連 活動報告, FIRM (2016).
9) MAQC Consortium: The MicroArray Quality Control (MAQC) project shows inter-and intraplatform reproducibility of gene expression measurements, Nature Biotechnology, Vol. 24, No. 9, pp. 1151-1161 (2006).
10) The External RNA Controls Consortium: The External RNA Controls Consortium: a progress report, Nature Methods, Vol. 2, No. 10, pp. 731-734 (2005).
11) 国立研究開発法人 産業技術総合研究所: 計量 標準総合センター 標準物質認証書 (2015). 12) H. Akiyama et al.: A set of external reference
controls/probes that enable quality assurance between different microarray platforms, Analytical
Bio-chemistry, Vol. 472, pp. 75-83 (2015).
13) M. Yoshioka, N. Kurosawa, M. Isobe: Target-selective joint polymerase chain reaction: A robust and rapid method for high-throughput production of recombinant monoclonal antibodies from single cells,
BMC Biotechnology, Vol. 11, pp. 75-86 (2011). 14) タカラバイオ(株)編: PCR実験の手引き, pp. 6–8 (2016). 15) 田村隆明編: 遺伝子工学実験ノート (上巻), 第 3版, pp. 175–176, 羊土社 (2010). 16) サーモフィッシャーサイエンティフィック(株) 編: リアルタイムPCRハンドブック, p. 8 (2016). 17) 清水宏: あたらしい皮膚科学, 第2版, pp. 3–9, 中山書店 (2011). 18) 川内康弘: 表皮角化細胞の分化と転写因子, 日 本皮膚科学会雑誌, Vol. 114, No. 2, pp.143–152 (2004). 19) 佐藤陽治: 再生・細胞医療の事業化におけるレ ギュラトリーサイエンスの役割, かながわ再 生・細胞医療産業化ネットワークキックオフ フォーラム, pp. 11–21 (2016). 20) 西田幸二: 次世代医療機器評価指標作成事業 再生医療審査WG報告書, p. 20 (2013).